بررسی کیفیت DNA استخراج شده
بررسی کیفیت DNA استخراج شده: پایه ای برای موفقیت در آزمایشهای ژنتیکی
بررسی کیفیت DNA استخراج شده : استخراج DNA اولین و یکی از حیاتیترین مراحل در اکثر آزمایشهای زیستشناسی مولکولی، از PCR و توالییابی (Sequencing) تا کلونینگ و مهندسی ژنتیک است. با این حال، خود فرآیند استخراج به تنهایی کافی نیست؛ ارزیابی دقیق کیفیت و کمیت DNA به دستآمده شرط لازم برای اطمینان از صحت و تکرارپذیری نتایج آزمایشهای بعدی است. DNA با کیفیت پایین میتواند منجر به شکست کامل آزمایش یا نتایج گمراهکننده و غیرقابل اعتماد شود. بنابراین، بررسی کیفیت DNA استخراج شده یک مرحله اجباری و غیرقابل چشمپوشی در هر آزمایشگاهی محسوب میشود.
بررس کیفیت DNA استخراج شده عمدتاً بر سه پایه استوار است:
-
خلوص (Purity)
-
کمیت (Quantity)
-
یکپارچگی (Integrity)
۱. خلوص (Purity): آیا DNA استخراج شده ما آلوده است؟
خلوص به میزان آلودگی نمونه DNA با سایر مولکولها اشاره دارد.آلایندهها شامل پروتئینها (به ویژه پروتئیناز K استفاده شده در مرحله لیز)، کربوهیدراتها، لیپیدها، نمکهای باقیمانده از بافرهای استخراج، و مهمتر از همه، RNA و فنول هستند. هر یک از این آلایندهها میتوانند در downstream applications اختلال ایجاد کنند.
روش اصلی ارزیابی خلوص: اسپکتروفتومتری (Spectrophotometry)
این روش با استفاده از دستگاه نانودراپ (NanoDrop) یا اسپکتروفتومترهای مشابه انجام میشود. اساس کار، جذب نور توسط مولکولها در طولموجهای خاص است.
-
DNA نور ماوراءبنفش (UV) را در طولموج ۲۶۰ نانومتر جذب میکند.
-
پروتئینها در طولموج ۲۸۰ نانومتر جذب بالایی دارند.
-
نمکها و فنول در طولموج ۲۳۰ نانومتر جذب دارند.
با اندازهگیری میزان جذب در این طولموجها، میتوانیم نسبتهای زیر را محاسبه کنیم که نشاندهنده خلوص نمونه هستند:
-
نسبت A260/A280: این معروفترین شاخص خلوص است.
-
یک نسبت ۱.۸ تا ۲.۰ برای DNA خالص ایدهآل در نظر گرفته میشود.
-
نسبت پایینتر از ۱.۸ معمولاً نشاندهنده آلودگی با پروتئین یا فنول است.
-
نسبت بالاتر از ۲.۰ ممکن است نشاندهنده آلودگی با RNA یا Degradation (تخریب) DNA باشد.
-
-
نسبت A260/A230: این شاخص خلوص در برابر آلایندههای آلی مانند نمکها، EDTA یا فنول حساس است.
-
یک نسبت مطلوب برای DNA خالص معمولاً در محدوده ۲.۰ تا ۲.۲ قرار دارد.
-
نسبت پایینتر از ۲.۰ (مثلاً ۱.۵ یا کمتر) به وضوح نشاندهنده آلودگی با نمکهای باقیمانده، کربوهیدراتها یا فنول است که میتوانند آنزیمهایی مانند Taq پلیمراز در PCR را مهار کنند.
-
محدودیت اسپکتروفتومتری: این روش نمیتواند بین DNA، RNA و نوکلئوتیدهای آزاد تفکیک قائل شود. همچنین برای نمونههای بسیار رقیق یا آلوده به مقادیر کمی از آلایندهها ممکن است دقت کافی نداشته باشد.
۲. کمیت (Quantity): چه مقدار DNA داریم؟
دانستن غلظت دقیق DNA برای بسیاری از آزمایشها حیاتی است. برای مثال، در PCR باید مقدار مشخصی از الگو (Template) استفاده شود تا واکنش بهینه باشد.
روشهای اندازهگیری کمیت:
-
اسپکتروفتومتری: سادهترین و سریعترین روش است. غلظت DNA (بر حسب نانوگرم بر میکرولیتر) با استفاده از میزان جذب در ۲۶۰ نانومتر محاسبه میشود (طبق قانون Beer-Lambert). یک واحد جذب (Absorbance) در A260 معادل تقریباً ۵۰ نانوگرم بر میکرولیتر DNA دو رشتهای (dsDNA) است.
-
فلورومتری (Fluorometry): این روش با استفاده از دستگاههایی مانند Qubit و dyes های فلورسنت اختصاصی (مانند Picogreen) که فقط به DNA دو رشتهای متصل میشوند، کار میکند. برخلاف نانودراپ، این dyes ها به RNA، نوکلئوتیدهای آزاد یا پروتئینها متصل نمیشوند. بنابراین، دقت فلورومتری به مراتب بالاتر از اسپکتروفتومتری است و برای نمونههای با غلظت بسیار کم یا نمونههای آلوده به RNA، روش ارجح محسوب میشود.
۳. یکپارچگی (Integrity): آیا مولکولهای DNA سالم و دستنخورده هستند؟
یکپارچگی به اندازه و سلامت مولکولهای DNA اشاره دارد. DNA استخراج شده ایدهآل باید به صورت رشتههای بلند و بدون شکستگی باشد. تخریب DNA (Degradation) میتواند به دلایلی مانند فعالیت نوکلئازهای داخلی، استخراج خشن یا نگهداری نامناسب رخ دهد.
روش اصلی ارزیابی یکپارچگی: الکتروفورز ژل آگارز (Agarose Gel Electrophoresis)
این روش قدرتمند و نسبتاً ساده، تصویری از کیفیت DNA ارائه میدهد.
-
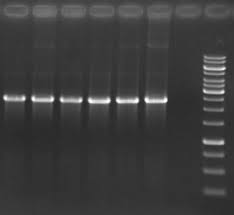
DNA ژنومی (gDNA) با کیفیت بالا: باید یک باند (band) متراکم، sharp و با وزن مولکولی بسیار بالا در نزدیک ترین چاهک (well) مشاهده شود. هیچ باندی که نشاندهنده تخریب باشد (شبیه یک smear از پایین به بالا) نباید دیده شود.
-
DNA تخریب شده (Degraded): به صورت یک smear (لکه) پیوسته از اندازههای مختلف از بالا به پایین ژل دیده میشود و باند متراکم اصلی وجود ندارد. چنین نمونهای برای بسیاری از آزمایشها، به خصوص توالییابی کل ژنوم یا library preparation، نامناسب است.
-
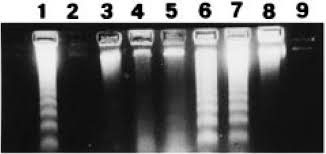
DNA تخریب شده (Degraded).به جای مشاهده باند شارپ اسمیر دیده می شود.
-
آلودگی با RNA: اگر در نمونه RNA آلوده وجود داشته باشد، یک باند یا smear روشن و منتشر در محدوده وزن مولکولی پایین (معمولاً زیر ۵۰۰ جفت باز) دیده میشود که نشاندهنده rRNA و tRNA است.
-
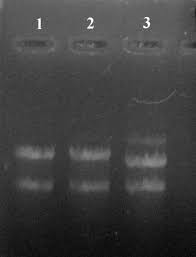
آلودگی با RNA, در این حالت علاوه بر باند اصلی باند دیگری هم در پایین باند اصلی دیده می شود.
برای بررسی دقیقتر یکپارچگی، به ویژه در کاربردهای حساس مانند microarray یا NGS، از دستگاه Bioanalyzer (شرکت Agilent) استفاده میشود. این دستگاه یک الکتروفورز مویینه خودکار انجام میدهد و یک الکترروفرام و عددی به نام عدد یکپارچگی RNA (RIN) یا معادل آن برای DNA (DIN) ارائه میکند که سلامت نمونه را به صورت کمی و بسیار دقیق نشان میدهد.
جمعبندی و انتخاب روش بر اساس کاربرد نهایی بررسی کیفیت DNA استخراج شده
هیچ روش واحدی به تنهایی نمیتواند تمام جنبههای کیفیت DNA را ارزیابی کند. یک رویکرد ترکیبی همیشه توصیه میشود:
-
غربالگری اولیه: از نانودراپ برای اندازهگیری سریع خلوص و کمیت تقریبی استفاده کنید. اگر نسبتهای A260/A280 و A260/A230 در محدوده قابل قبول بودند، به مرحله بعد بروید.
-
تأیید کمیت دقیق: برای آزمایشهای حساس، غلظت را با Qubit اندازهگیری کنید تا از دقت بالاتر اطمینان حاصل شود.
-
بررسی سلامت نمونه: حتماً الکتروفورز ژل انجام دهید تا از عدم تخریب DNA و نبود آلودگی RNA مطمئن شوید.
نتیجهگیری نهایی:
بررسی کیفیت DNA استخراج شده یک گزینه لوکس نیست، بلکه یک ضرورت مطلق است. سرمایهگذاری اندک در زمان و مواد برای این ارزیابی، از هدر رفتن منابع بسیار بیشتر (مانند reagents گرانقیمت، زمان نیروی انسانی و نمونههای ارزشمند) در مراحل بعدی جلوگیری میکند و تضمین میکند که دادههای تولید شده قابل اعتماد، دقیق و قابل انتشار هستند. یک نمونه با کیفیت خوب، نصف راه موفقیت در هر آزمایش مولکولی است.
شرکت پویا ژن آزما مفتخر است که در تولید کیت های استخراج DNA و خالص سازی، به صورت کاملا داخلی و با قیمت مناسب در کنار علکرد بی رقیب خدمت رسان دانشجویان و محققین محترم باشد.



